SEPTEMBER 27, 2023 | SEQUENCING METHODS
マルチオームへ: 1回のRevio SMRT Cellで得られた4つの高品質な'ome'
< Return to Japan blog | < Return to Main blog

マーベルのスーパーヒーローが多元宇宙を旅して窮地を救うように、ゲノム研究者は生物学という困難な多元宇宙を旅する我々のスーパーヒーローです。ゲノム配列とそのエピジェネティックな制御、そしてそれらが転写産物の発現やスプライシングに及ぼす複合的な影響との間の複雑でダイナミックな相互作用は、生物学を理解する上で基本的なものであり、それらの異常が病気を引き起こします。マルチオミクス・アプローチは、ゲノムからトランスクリプトームへの根本的な情報のつながりや、その流れをよりよく理解することを可能にします。現在では生物医学研究において当たり前のものとなっていますが、マルチオミクス研究は、複数のアッセイ、シーケンスラン、テクノロジーを必要とし、別々のデータセットを組み合わせる必要があるため、コストと労力がかかるという問題があります。
このブログでは、マルチオミクス研究の新たなスーパーパワーを紹介します。ワシントン大学のAndrew Stergachis博士とMitchell Vollger博士が率いる共同研究による新しいプレプリント『Synchronized long-read genome, methylome, epigenome, and transcriptome for resolving a Mendelian condition』では、1回のRevio SMRT Cellシーケンスから、ゲノム、CpGメチローム、クロマチンエピゲノーム、トランスクリプトームという4つの高品質でハプロタイプ解像度の‘ome’を同時に生成したことが述べられています。
この新しい能力は、3つの先端技術を組み合わせることによって達成されます。第一に、PacBio Revioシステムの高いスループットを活用し、高品質のロングリードHiFiシーケンスデータをスケールアップして取得します。第二に、ワシントン大学の研究者らは、ハプロタイプ解像度の1分子クロマチンアクセシビリティ、ヌクレオソームの位置、転写因子の占有パターンを正確に組み立てるために、クロマチンファイバーseqデータの機械学習アプローチを開発しました(Stergachis et al. )。第三に、MAS-Seq法(Al’Khafaji et al. 2023)のライブラリープロトコルを応用して、完全長cDNA分子を連結し、完全長RNAシーケンスのスループットを向上させました。連結したcDNAライブラリーは、標準的なHiFi WGSライブラリーと同様のフラグメントサイズをターゲットとしており、Revioシステムでの1回のHiFiシーケンスランで、Fiber-Seq処理したgDNAライブラリーと一致するcDNAライブラリーを組み合わせることが可能になっています。
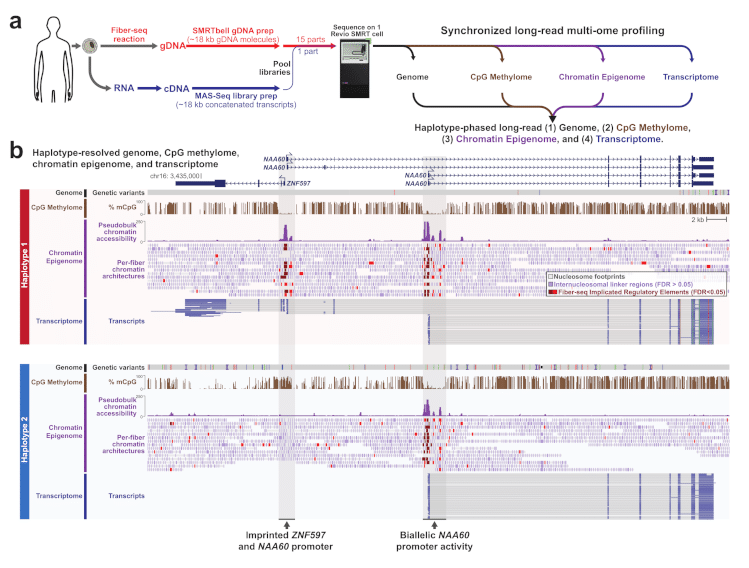
Vollger et al.の図1:ロングリードゲノム、メチローム、エピゲノム、トランスクリプトームシーケンスを同期化
a, 同期マルチオームプロファイリングの実験と計算ワークフローを説明する概略図。具体的には、細胞はFiber-seq反応後、ゲノムDNA抽出とSMRTbellライブラリー調製を行い、並行して細胞からRNA抽出後、相補的DNA(cDNA)合成とMAS-seqライブラリー調製を行う。その後、2つのライブラリーを混合し、1回のシーケンスランで同時にシーケンスすることで、サンプルからゲノム、CpGメチローム、クロマチンエピゲノム、トランスクリプトームを同時に検出することができる。b,既知のインプリンティング遺伝子座におけるGM12878細胞からのハプロタイプ分解されたゲノム、CpGメチローム、クロマチンエピゲノム、トランスクリプトームを示すゲノム領域の例。
まず、この同期マルチオームアッセイの性能を、既知の検証済み情報をもつGenome-in-a-Bottle(GIAB)サンプルに対してベンチマークし、Fiber-seqデータが、すべてのバリアントタイプ(SNV、インデル、SV)、CpGメチル化検出、および二倍体de novoゲノムアセンブリの品質(ハプロタイプコンティグN50が20 Mb以上、コンセンサス精度QV 50)を高精度に保持することを実証しました。加えて同じRevio SMRTセルでシーケンスされデコンカテネーションされたcDNAリードは、これらのGIABサンプルの全長RNA転写データと一致しました。
次に、このマルチオームアプローチを、原因不明の発達遅滞、多発性小耳症、感音性難聴、および両側網膜芽細胞腫を有するUDN(Undiagnosed Diseases Network)参加者の検体に適用しました。1個のRevio SMRTCellから得られたマルチオームデータは、それぞれの’ome’が、この個体の複数の疾患症状の分子的基盤の説明に貢献し、多数の表現型に4つの異なる遺伝子が関与していることを明らかにしました。

Vollger et al.の表2: UDN参加者に同定された分子変異体およびその同定に必要な‘ome’の概要
これらの結果は、原因不明の希少疾患の根底にあるメカニズムを解明するための、統合された単一技術によるロングリードマルチオームプロファイリングの強力な有用性を示しています。ここでは、これまで原因不明であった疾患を持つヒトゲノムについて実証していますが、この新しいシンクロマルチオームパラダイムは、分子構造のより深い理解が求められるあらゆる生物学的サンプルに適用することができます。つまり、このパラダイムは、がん、動植物ゲノム、集団ゲノム、その他多くの分野で大きな可能性を秘めています。私たちは、生物学的サンプルの多くの側面を同時に解明することで、これらすべての分野でどのような発見と理解が待っているのか、つまり、あらゆる場所であらゆることを一度に知ることができるのか、をとても楽しみにしています。
PacBioに問い合わせる